I. PENDAHULUAN
a.
Latar Belakang
DNA merupakan molekul
yang penting bagi makhluk hidup. DNA merupakan materi genetik yang mampu
mengkodekan perintah-perintah untuk mengatur perkembangan maupun respon-respon
biologis tertentu. Isolasi DNA merupakan langkah yang sangat penting dalam
mempelajari DNA. Terdapat beragam metode yang dapat digunakan dalam isolasi
DNA. Metode tersebut seringkali harus disesuaikan dengan tipe sel yang akan
kita isolasi DNAnya. Salah satu metode yang dapat digunakan dalam isolasi DNA
tumbuhan adalah metode CTAB. CTAB merupakan sejenis detergen yang mampu
melisiskan sel. Selain CTAB, diperlukan reagen-reagen lain dalam isolasi DNA
mengingat tumbuhan memiliki struktur serta muatan sel yang sangat kompleks dan
berpotensi mengkontaminasi hasil isolasi DNA.
Isolasi DNA merupakan
langkah awal yang biasa dilakukan dalam studi molekuler keragaman genetik. DNA
dari makhluk hidup akan berbeda satu sama lain. Hal ini mengakibatkan keragaman
yang tinggi pada makhluk hidup. Salah satu metode yang dapat digunakan adalah
RAPD. RAPD atau Random Amplified
Polymorphic DNA merupakan metode
amplifikasi menggunakan primer oligonukleotida yang bersifat tidak spesifik.
Metode ini dapat digunakan sebagai metode karakterisasi makhluk hidup.
Elektroforesis
dengan Agarose merupakan metode standar untuk memisahkan, mengidentifikasi,
mengkarakterisasi dan purifikasi dari molekul DNA. Elektroforesis
memanfaatkan adanya muatan dalam suatu molekul untuk memisahkannya dalam
lingkungan medan listrik.
b.
Permasalahan
1.
Bagaimana cara mengisolasi
DNA dari jaringan tumbuhan dengan metode CTAB?
2.
Bagaimana cara melakukan
amplifikasi DNA menggunakan RAPD?
3.
Bagaimana cara melakukan
uji kualitas DNA menggunakan elektroforesis?
c. Tujuan
1.
Melakukan Isolasi DNA
dari jaringan tumbuhan dengan metode CTAB.
2.
Melakukan amplifikasi
DNA menggunakan RAPD.
3.
Melakukan uji kualitas
DNA menggunakan elektroforesis.
II.
TINJAUAN PUSTAKA
a.
Landasan Teori
Ekstraksi biomolekul,
termasuk DNA merupakan salah satu metode krusial dalam biologi molekuler. Pada
umumnya, isolasi asam nukleat memerlukan empat tahapan yaitu penghancuran sel
yang efektif, denaturasi kompleks protein, inaktifasi enzim nuklease, serta
purifikasi sehingga tidak terdapat kontaminan (Chee Tan, 2009). Asam nukleat
target harus bebas dari kontaminan seperti protein, karbohidrat, lipid, maupun asam
nukleat lain. Purifikasi dari protein merupakan langkah penting dalam isolasi
DNA. Dalam ekstraksi DNA tanaman, tahapan yang diperlukan diawal adalah
menghaluskan jaringan sampel. Pada tahapan ini biasanya digunakan liquid
nitrogen untuk mempermudah penghancuran. Pada tahapan ini, dinding sel
diharapkan dapat hancur. Namun demikian, perlu ada upaya inaktifasi enzim dan
material kimia seluler agar target DNA tidak terdegradasi.
1. Tahapan
isolasi DNA dengan CTAB
Sel
tanaman dapat dilisiskan menggunakan CTAB. Cetyltrimethylammonium bromid (CTAB)
merupakan detergen yang mampu mempresipitasikan asam nukleat serta polisakarid
yang bersifat asam dari larutan dengan kekuatan ionik rendah. Pada kondisi kekuatan
ionik yang tinggi, CTAB tidak mampu mempresipitasi asam nukleat dan membentuk
kompleks dengan protein (Chee Tan, 2009). Oleh sebab itu, CTAB dapat berguna
untuk purifikasi asam nukleat untuk sel dengan kuantitas polisakarida tinggi
seperti tumbuhan dan bakteri tertentu. Methode CTAB juga menggunakan pelarut
organik dan alkohol dalam tahap presipitasi. Partikel yang tidak larut akan
dihilangkan menggunakan sentrifugasi.
Terdapat
beberapa tahapan penting dalam isolasi DNA menggunakan CTAB sebagai berikut (M.
Somma, 2001).
1. Melisiskan
sel
Tahapan
pertama dalam isolasi DNA adalah melisiskan sel. pada tahapan ini, sampel yang
homogen diberi dengan buffer EDTA dan CTAB. Membran sel memiliki komponen lipid
dan protein yang saling terhubung dengan ikatan nonkovalen. Molekul lipid
memiliki kepala hidrofobik dan ekor hidrofilik. CTAB akan menangkap lipid dan
protein serta membiarkan DNA genom. Garam spesifik yang ditambahkan kemudian
akan mengikatkan DNA kepada detergen. EDTA merupakan buffer yang mampu mengikat
magnesium yang merupakan kofaktor DNAse. Dengan adanya pengikatan ion magnesium
oleh EDTA, maka DNA akan terhindar dari degredasi. Tris HCL merupakan buffer pH
yang penting untuk menyediakan lingkungan bagi DNA agar tidak terdegredasi.
2. Ekstraksi
Pada
tahapan ini polisakarida, komponen fenolik serta protein dan lisat lainnya
dilarutkan ke dalam fase larutan dan dipisahkan dari kompleks CTAB dan asam
nukleat. Pada konsentrasi garam yang rendah, kontaminan dari kompleks asam
nukleat tidak dapat larut dan dapat dipisahkan dengan ekstraksi dengan
kloroform. Kloroform mendenaturasi protein dan memfasilitasi pemisahan fase
organik dan aqueous. Fase aqueous biasanya terdapat diatas, namun demikian,
karena konsentrasi garam tertentu maka akan berada di bawah. Asam nukleat akan
berada pada fase organik. Ekstraksi menggunakan kloroform dapat diulang sesuai
kebutuhan untuk meningkatkan kemurnian.
3. Presipitasi.
Pada
tahap akhir, asam nukleat dibebaskan dari detergen. Pada tahapan ini
makaterlebih dahulu dilakukan presipitasi dengan menggunakan campuran CTAB dan
garam. Garam diperlukan untuk mempresipitasi asam nukleat. Dengan adanya
kondisi buffer dan garam, maka detergen akan dapat dipisahkan dari molekul DNA.
Setelah perlakuan ini, maka dapat dilakukan purifikasi dengan etanol 70% untuk
membersihkan asam nukleat dari sisa garam yang ada.
2. Uji
kuantitas DNA dengan spektrofotometri
Konsentrasi
DNA dapat diketahui dengan menggunakan spektrofotometri. Dengan menggunakan
spektrofotometer, protein menyerap gelombang 280 nm, DNA 260 nm, sedangkan RNA
280 nm. Rasio kemurnian yang baik adalah rasio kemurnian diatas 1,8 dan
mendekati 2. Absorpsi pada panjang gelombang 230 nm menunjukkan adanya
kontaminasi dengan materi seperti karbohidrat, peptida, fenol maupun komponen
aromatik lainnya. Spektrofotometer mentransmisikan cahaya melalui larutan untuk
mengetahui konsentrasi zat terlarut dalam larutan. Nilai absorbansi yang
ditunjukkan oleh spektrofotometer akan berbanding lurus dengan konsentrasi yang
dimiliki oleh zat terlarut.
3. Uji
kualitas DNA dengan elektroforesis
Elektroforesis agarose
merupakan metode yang sering digunkan untuk memisahkan protein, DNA, maupun
RNA. Asam nukleat merupakan molekul dengan muatan negatif sehingga dapat
bergerak ke kutub positif di bawah medan listrik (Yilmaz et al, 2015). Dalam
migrasinya, semakin kecil berat molekul maka akan semakin cepat migrasi yang
terjadi. Molekul-molekul dengan ukuran yang sama pada akhirnya akan membentuk
band yang sama. Dalam visualisasi molekul asam nukleat, biasanya diperlukan
pewarna berupa etidium bromida maupun SYBR green yang mampu berpendar dengan
adanya paparan terhadap sinar UV 300 nm.
4.
RAPD (Random Amplified Polymorphic DNA)
RAPD adalah teknik PCR
untuk mengidentifikasi variasi genetik. RAPD menggunakan primer pendek sehingga
bersifat tidak spesifik (Kumari & Thakur, 2014). Dengan menggunakan teknik
ini, primer akan menyeleksi band yang
akan terbentuk dengan sangat spesifik. RAPD menggunakan primer berupa sekuens
basa oligonukleotida yang pendek untuk mengamplifikasi genom DNA dengan
temperatur annealing yang rendah
menggunakan PCR. Hasil amplifikasi kemudian dipisahkan dengan menggunakan gel
elektroforesis. Dalam aplifikasi, sama seperti halnya PCR pada umumnya terdapat
3 tahapan yang terjadi. tahapan tersebut adalah sebagai berikut.
1. Denaturasi
Denaturasi
merupakan proses pemisahan untai ganda DNA. pada tahap ini biasanya diperlukan
suhu yang cukup tinggi untuk dapat melepaskan ikatan hidrogen antara dua untai
DNA.
2. Penempelan.
Pada
tahapan ini biasanya digunakan suhu yang relatif rendah. Dalam tahap ini,
primer akan menempel pada sekuens Dna spesifik yang komplemen dengan sekuens
yang dimiliki primer.
3. Pemanjangan
Tahapan
ini merupakan tahapan pembentukan sekuens DNA baru. Proses ini dibantu oleh
adanya enzim Taq polimerase yang mampu bekerja pada suhu yang tinggi.
b. Hipotesis
1.
Isolasi DNA dari daun
melon dapat dilakukan dengan metode CTAB.
2.
Amplifikasi DNA
menggunakan thermo cycler menghasilkan amplikon.
3.
Kualitas DNA dapat
dilihat menggunakan elektroforesis.
III.
METODOLOGI PENELITIAN
a. Alat
Alat
yang digunakan dalam isolasi DNA adalah mikropipet beserta tip, microtube 1,5
ml dan 0,5 ml, alumunium foil,vorteks, oven, waterbath, pendingin, serta
permanent marker. Alat yang digunakan dalam uji kualitas DNA dengan
elektroforesis adalah mikropipet dan tip, erlenmeyer, microwave, glass plate,
serta set elektroforator. Alat yang digunakan dalam amplifikasi DNA adalah
mesin thermo cycler serta microtube.
b. Bahan
Bahan
yang digunakan dalam isolasi DNA adalah daun melon GMP, HIK, MG3, MG1, TCP, dan
MLN masing-masing sebanyak 0,2 gram, CTAB, PVP, merkaptoetanol, CIA,
isopropanol, etanol absolut, etanol 70%, buffer TE. Bahan yang digunakan untuk
menguji kualitas DNA dengan elektroforesis adalah agarosa, buffer TAE 1X dan
1/2X, flourosafe, loadingdye. Bahan yang digunakan dalam amplifikasi DNA adalah
mix PCR Kapa, primer OP7, dH2O, serta sampel DNA.
c. Cara
kerja
1. Isolasi
DNA
Sampel
daun terlebih dahulu ditimbang sebanyak 0,2 gram dan dibalut dengan alumunium
foil. Sampel kemudian digerus menggunakan mortar hingga halus dan homogen.
Sampel tersebut kemudian ditambahkan 300 µl CTAB 2% serta 0,02 gram PVP lalu
dimasukkan ke dalam microtube 1,5 ml. Lalu tambahkan lagi CTAB 2% sebanyak 400
µl. Selanjutnya, ke dalam tube ditambahkan 75 µl merkaptoetanol dan diinversi 7
kali. Sampel dalam tube kemudian diinkubasi pada suhu 65oC selama 10
menit. Setelah 10 menit, sampel ditambahkan dengan CIA dingin sebanyak 600 µl
dan dihomogenasi selama 20 menit. Selanjutnya dilakukan sentrifugasi dengan
kecepatan 13.000 rpm selama 10 menit. Supernatan yang dihasilkan kemudian
dipindah ke tube baru dan ditambahkan dengan isopropanol sesuai dengan volume
supernatan yang diambil (1:1). Selanjutnya dilakukan inversi sebanyak 14 kali
dan diinkubasi selama 10 menit pada suhu -20oC. Setelah inkubasi
dilakukan sentrifugasi dengan kecepatan 13.000 rpm selama 10 menit. Supernatan
yang dihasilkan selanjutnya dibuang dan dikering-anginkan sebentar. Selanjutnya
ditambahkan 100 µl etanol absolut dan kembali dilakukan sentrifugasi dengan
kecepatan 13.000 rp, selama 10 menit. Selanjutnya supernatan yang dihasilkan
dibuang lalu ke dalam tube ditambahkan alkohol 70%. Setelah ditambahkan alkohol
70% selanjutnya kembali dilakukan sentrifugasi selama 5 menit dengan kecepatan
13.000 rpm. Supernatan dibuang dan pelet dikeringkan untuk menghilangkan sisa
alkohol 70%. Selanjutnya dilakukan penambahan buffer TE sebanyak 50 µl dan
disimpan pada suhu -20oC.
2. Amplifikasi
DNA dengan teknik RAPD menggunakan thermo cycler.
Sampel
yang akan diloading dipersiapkan terlebih dahulu. Satu tube sampel terdiri dari
12,5 µl mix Kapa, 1 µl MgCl2 , 1,25 µl primer forward, 1,25 µl
primer reverse, 5 µl DNA template, serta 4 µl ddH2O. Tube berisi
campuran tersebut kemudian ditata di dalam thermo cycler dan diatur untuk suhu
predenaturasi 95oC selama 5 menit, suhu annealing 40oC
selama 15 detik, suhu extension 72oC selama 20 detik, serta suhu
final extension sebesar 72oC selama 5 menit dengan jumlah ulangan
siklus sebanyak 35 kali. Setelah proses amplifikasi selesai kemudian sampel
dapat disimpan pada suhu sekitar 4oC hingga 12oC serta
selanjutnya dilakukan uji kualitas dengan menggunakan elektroforesis.
3. Uji
kualitas DNA menggunakan elektroforesis
Pada
uji kualitas DNA genom menggunakan elektroforesis, terlebih dahulu disiapkan
agarose dengan konsentrasi 1%. Sebanyak 0,4 gram agarose dimasukkan ke dalam
gelas ukur dan ditambahkan dengan 40 ml TAE 1X lalu digojok hingga homogen.
Sedangkan untuk uji kualias produk PCR digunakan 2% agarose dengan melarutkan
0,8 gram agarose ke dalam 40 ml TAE. Larutan agarosa kemudian dipanaskan dalam
microwave selama 50 detik. Setelah agak dingin, ditambahkan dengan flourosafe
sebanyak 2 µl dan digojok hingga homogen. Gel kemudian dituangkan dalam cetakan
dan didiamkan hingga memadat lalu setelah memadat sisit dilepas kembali. Gel
dipindahkan ke dalam tangki elektroforesis yang berisi buffer TAE 1/2x.
Sembari
menyiapkan set elektroforesis, sampel yang akan diloading ke dalam sumuran
dipersiapkan terlebih dahulu. Untuk sampel genom, diambil sebanyak 5 µl sampel
serta ditambahkan dengan loading dye sebanyak 1 5 µl. Setelah sampel siap di
dalam sumuran, elektroforator dihubungkan dengan sumber listrik dan di atur
pada tegangan 100V selama 30 menit. Setelah selesai, hasilnya diamati
menggunakan sinar UV.
IV.
HASIL DAN PEMBAHASAN
Pada praktikum ini
telah dilakukan praktikum isolasi DNA, uji kualitas DNA dengan elektroforesis,
serta amplifikasi DNA dengan thermocycler.
Kegiatan pertama yang
dilakukan adalah isolasi DNA. Dalam praktikum ini dilakukan isolasi DNA
menggunakan CTAB. Langkah pertama yang dilakukan adalah menimbang sampel
sebanyak 0,2 gram. Sampel yang digunakan adalah sampel daun melon melon GMP,
HIK, MG3, MG1, TCP, dan MLN. Sampel tersebut dipilih yang tidak terlalu tua
sehingga tidak memiliki kandungan metabolit sekunder yang terlalu banyak yang
dapat mengganggu kemurnian DNA nantinya.
Tulang daun pada
tanaman merupakan bagian yang sedapat mungkin dihilangkan karena akan
mengganggu proses penghancuran sel. selain itu, tulang daun merupaka
representasi berkas pengangkut sehingga tipe sel yang ada di dalamnya cukup
banyak sel mati atau tidak memiliki protoplas. Oleh sebab itu, keberadaan
tulang daun sebaiknya dihindari. Tulang daun juga merupakan sarana transportasi
sehingga di dalam tulang daun kemungkinan terdapat berbagai macam substansi
yang bukan merupakan target isolasi, yaitu DNA. Sampel yang sudah ditimbang
disimpan pada alumunium foil dan disimpan pada suhu rendah agar metabolisme
pada sel-sel sampel tidak berlanjut. Suhu yang rendah mampu menurunkan
aktifitas enzim sehingga reaksi-reaksi dalam sel menjadi lebih lambat. Keadaan
ini penting sehingga keadaan sel dalam jaringan tetap baik selama penyimpanan
sampel.
Setelah sampel selesai
disiapkan, langkah selanjutnya adalah menghaluskan sampel dengan mortar.
Langkah ini bertujuan untuk menghancurkan dinding sel. Pada langkah ini
ditambahkan sebanyak 300 µl CTAB dengan suhu 60oC serta o,o2 gram
PVP. Penambahan CTAB bertujuan untuk melisiskan membran sel. selain itu,
perbedaan suhu penyimpanan serta suhu CTAB yang ditambahkan diharapkan dapat
memberikan shock temperatur bagi
membran sel sehingga mudah dilisiskan. CTAB merupakan sejenis detergen yang
mampu melisiskan dinding sel. Sedangkan penambahan PVP bertujuan untuk
menghilangkan polifenol yang merupakan agen pengoksidasi yang mampu menurunkan
kemurnian DNA hasil ekstraksi ekstraksi (Khanuja et al, 1999). Selanjutnya,
sampel yang sudah homogen serta telah ditambahkan CTAB dan PVP dipindahkan ke
dalam microtube 1,5 ml serta ditambahkan kembali 400 µl CTAB dan 75 µl
merkaptoetanol.
Fungsi dari
merkaptoetanol adalah untuk mendenaturasi protein (Miguel Dita & Martinez
de la Parte, 2014). Setelah penambahan CTAB dan merkaptoetanol dilakukan
inversi agar larutan menjadi homogen. Homogenasi ini penting agar kontak buffer
dengan substansi yang akan dilisiskan lebih maksimal. Selanjutnya, sampel
diinkubasi pada suhu 65oC selama 10 menit untuk denaturasi protein
lebih lanjut. Suhu yang lebih tinggi akan mampu mempercepat proses denaturasi
protein.
Setelah inkubasi
selesai, sampel kemudian ditambahkan dengan CIA 600 µl dingin dan dilakukan
homogenasi selama 20 menit. Penggunaan CIA berperan dalam purifikasi DNA.
Kloroform dalam CIA berperan dalam mendenaturasi protein dan menghilangkan
lipid. Isoamil alkohol berperan dalam pemisahan fase, penrunan jumlah material
dalam fase aqueous dan interfase organik, serta mengurangi terbentuknya buih.
CIA diberikan dalam keadaan dingin sehingga kerapatan molekul protein dan
debris lainnya tinggi sehingga akan berada di bagian bawah tabung saat
dilakukan sentrifugasi pada langkah berikutnya. Pada tahapan ini materi dengan
massa jenis dan kelarutan yang sama akan berada dalam fase yang sama sehingga
membentuk gradien fase. Sentrifugasi dilakukan dengan kecepatan 13.000 rpm
selama 10 menit.
Setelah sentrifugasi,
supernatan dipindahkan ke dalam tube baru menggunakan mikropippet. Kemampuan
pipeting yang baik akan membantu keberhasilan isolasi DNA. Pengambilan
supernatan harus dilakukan dengan sangat hati-hati agar debris tidak kembali
bercampur ke dalam supernatan dan kemudian mengurangi kemurnian isolat DNA.
Selanjutnya dilakukan
penambahan isopropanol sebanyak supernatan yang diambil sebelumnya. Fungsi dari
isopropanol adalah untuk mempresipitasi DNA. Isopropanol akan mengakibatkan
kerapatan dsDNa meningkat sehingga mudah terpresipitasi. Isopropanol memiliki
konstanta dielektrik yang lebih kecil dari air. Isopropanol bersifat lebih
tidak polar daripada etanol absolut. Hal ini mengakibatkan kemungkinan adanya
garam tertentu yang tidak dapat larut saat dilakukan presipitasi dengan
isopropanol.
Setelah ditambahkan
isopropanol dilakukan inversi sebanyak 14 kali dan inkubasi selama 10 menit
pada suhu -20oC. Inkubasi pada suhu rendah juga akan meningkatkan
kerapatan molekul DNA. Dengan demikian, pada tahap selanjutnya yaitu
sentrifugasi selama 10 menit dengan kecepatan 13.000 rpm diperoleh pelet DNA di
dasar tube. Sampel kemudian dikeringanginkan sebentar.
Setelah presipitasi
menggunakan isopropanol, dilakukan kembali presipitasi menggunakan etanol
absolut. Etanol absolut bersifat lebih polar sehingga lebih mampu melarutkan
garam-garam yang mungkin terdapat dalam sampel DNA. Setelah penambahan etanol
absolut kemudian dilakukan sentrifugasi 13.000 rpm selama 10 menit. Superatan
yang dihasilkan kemudian dibuang. Selanjutnya ditambahkan etanol 70% sebagai
tahap pencucian. Setelah itu, kembali dilakukan sentrifugasi dengan kecepatan
13.000 rpm selama 5 menit. setelah selesai, supernatan dibuang dan sampel
dikeringkan. Pengeringan tidak boleh dilakukan berlebihan karena dapat
menurunkan kelarutan DNA pada buffer penyimpan selanjutnya. Setelah DNA kering,
kemudian ditambahkan TE sebanyak 50 µl sehingga DNA bebas dari kemungkinan
adanya degradasi serta lebih mampu bertahan dalam masa penyimpanan.
Setelah berhasil
diisolasi, genom kemudian dianalisis menggunakan spektrofotometer. Hasilnya
adalah sebagai berikut.
Tabel 1. Hasil
Spektrofotometri
|
Sampel
|
Konsentrasi (ng/
µl) |
A
230 |
A260 |
A280 |
A260/280 |
A260/230 |
|
GMP |
2628
|
28,26 |
54,64 |
27,58 |
2,061 |
2,008 |
|
HIK |
1004 |
10,64 |
21,12 |
10,71 |
2,075 |
2,091 |
|
MG3 |
1200 |
14,19 |
25,51 |
13,57 |
1,990 |
1,893 |
|
MG1 |
3248 |
34,32 |
67,04 |
34,26 |
2,019 |
2,015 |
|
TCP |
1906 |
19,00 |
39,08 |
19,00 |
2,113 |
2,113 |
|
MLN |
4033 |
41,10 |
81,94 |
41,68 |
1,997 |
2,026 |
Nilai 260/280 idealnya
berada pada kisaran 1,8 hingga 2. Pada praktikum ini, diketahui nilai tersebut
pada masing-masing sampel yang dianalisis memiliki nilai melebihi dua. Hal ini
mengindikasikan adanya kontaminasi substansi lain. selain itu, nilai absorbansi
230 juga tergolong tinggi. Hal ini menunjukkan adanya kemungkinan adanya
kontaminasi senyawa seperti karbohidrat,
peptida, fenol maupun komponen aromatik lainnya.
Pada kegiatan
selanjutnya, dilakukan amplifikasi DNA. Amplifikasi DNA dilakukan untuk
menghasilkan produk berupa amplikon yang merupakan sekuens DNA tertentu. Pada
praktikum ini, sampel yang akan diloading dipersiapkan terlebih dahulu. Satu
tube sampel terdiri dari 12,5 µl mix Kapa, 1 µl MgCl2 , 1,25 µl
primer forward, 1,25 µl primer reverse, 5 µl DNA template, serta 4 µl ddH2O.
Primer yang digunakan dalam praktikum ini adalah primer RAPD OPA-07 yang
memiliki sekuens 5’-GAACGGGTG-3’.
Tube berisi campuran
tersebut kemudian ditata di dalam thermo cycler dan diatur untuk suhu
predenaturasi 95oC selama 5 menit, suhu annealing 40oC
selama 15 detik, suhu extension 72oC selama 20 detik, serta suhu
final extension sebesar 72oC selama 5 menit dengan jumlah ulangan
siklus sebanyak 35 kali. Dalam RAPD ini, sekuens primer oligonukleotida akan
berperan sebagai primer forward sekaligus reverse. Primer ini akan menempel
pada banyak lokasi pada genom. Daerah antara primer forward dan reverse yang
berlawan arah dan berdekatan kemudian akan mampu mensintesis sekuens DNA. Setelah
proses amplifikasi selesai kemudian sampel diuji kualitasya dengan menggunakan
elektroforesis. Hasil analisis elektroforesis yang diperoleh adalah sebagai
berikut.
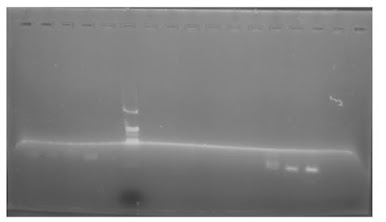
Gambar
1. Hasil Elektroforesis DNA Genom
Gambar
2. Hasil Elektroforesis DNA Produk RAPD
Hasil elektroforesis
DNA genom (Gambar 1) pada keenam sampel daun melon menunjukkan adanya smear yang cukup terang. Hal ini
menunjukkan adanya kontaminasi pada hasil isolasi DNA yang didapatkan. Pada
saat isolasi DNA di awal, diketahui bahwa pelet yang dihasilkan cenderung
berwarna putih dan kecoklatan. Hal ini menunjukkan kemungkinan adanya polisakarida
serta senyawa fenol. Nilai absorbansi 320 nm serta 230 nm juga cukup tinggi.
Apabila demikian, maka sebaiknya dilakukan pengulangan tahapan pemberian CTAB
dan PVP serta merkaptoetanol. Hasil isolasi DNA yang baik seharusnya
menunjukkan warna bening saat diamati secara langsung sebab DNA hanya mampu
menyerap sinar UV dengan panjang gelombang 260 nm sehingga tidak tampak dengan
mata secara langsung yang hanya mampu menangkap sinar tampak (400-750 nm). Hasil
ini menunjukkan bahwa tingginya kuantitas yang ditunjukkan oleh
spektrofotometer belum menjamin tingginya kualitas setelah diuji dengan
elektroforesis.
Pada Gambar 2,
diketahui bahwa PCR produk tidak dapat dilihat dengan jelas. Dengan menggunakan
primer OPA-07 seharusnya didapatkan 7 fragmen dengan perbandingan 3 fragmen DNA
polimorfik dan 4 fragmen DNA monomorfik dengan ukuran DNA sekitar 200-1000 bp
(Latifah, 2016). Namun demikian, band tersebut
tidak dapat teramati. Hal ini dimungkinkan oleh adanya beberapa faktor seperti
pembuatan gel yang belum rata kelarutannya, ketidakberhasilan PCR karena
terdapat gelembung pada mix sampel, maupun kesalahan yang mungkin terjadi
karena alat yang elektroforator yang tidak stabil.
V.
KESIMPULAN
1. Isolasi
DNA dari jaringan daun tanaman melon dapat dilakukan dengan menggunakan metode
CTAB.
2.
Amplifikasi DNA dapat
dilakukan dengan primer oligonukleotida yang bersifat tidak spesifik
menggunakan teknik RAPD.
3.
Elektroforesis dapat
digunakan untuk menguji kualitas DNA hasil isolasi maupun amplifikasi.
VI.
DAFTAR PUSTAKA
Chee
Tan, Siun, Beow Chin Yiap. 2009. DNA, RNA, and Proein Extraction: The Past and
The Present. Journal of Biomedicine and
Biotechnology Vol 2009 Article ID
574398
Khanuja, Suman P.S., Ajit K Shasany, MP
Darokar, Sushil Kumar. 1999. Rapid Isolation of DNA from Dry and Fresh Samples
of Plants Producing Large Amounts of Secondary Metabolites and Essential Oils. Plant Molecular Biology
Reporter 17: 1–7, 1999
Latifah,
Yulia Wahyu. 2016. Kestabilan Karakter
Fenotip dan Molekuler Melon (Curcumis melo L) Hasil Segregasi dan Seleksi
Populasi. Skripsi. Universitas Gadjah Mada
M.
Somma. The Analysis of Food Samples for
the Presence of Genetically Modified Organism Sesion 4: Extraction and
Purification of DNA. Online
http://gmo-crl.jrc.ec.europa.eu/capacitybuilding/manuals/Manual%20EN/Session04.pdf
diakses Sabtu, 20 Mei 2017
Miguel
Dita & Einar Martinez de la Parte. 2014. Basics Aspect of Molecular Biologi and DNA Extraction. Online
http://www.fao.org diakses Sabtu, 20 Mei
2017
Nandani
Kumari and Saroj Kumar Thakur. 2014. Randomly Amplified Polymorphic DNA-A Brief
Review. American Journal of Animal and
Veterinary Sciences 9 (1): 6-13
Porebski, S., Bailey, L.G. & Baum,
B.R. Modification of a CTAB DNA extraction protocol for plants containing high
polysaccharide and polyphenol components. Plant Mol Biol Rep (1997) 15: 8. doi:10.1007/BF02772108
Yilmaz,
Muhittin, Cem, Ozic, Ilhami Gok. 2015. Principles of Nucleic Acid Separation by
Agarose Gel Electrophoresis. Intech
VII.
LAMPIRAN